<dplyr 패키지란?>
dplyr 패키지는 데이터 프레임에 대한 일반적인 데이터 전처리 및 분석을 돕는 문법입니다.
즉, 기존 데이터셋을 특정 유형의 분석, 또는 데이터 시각화에 더 적합한 형식으로 변환하기 위한 동사(verbs)를 제공하는 R의 가장 대표적인 패키지이죠.
<dplyr 패키지 다운로드>
dplyr 패키지를 다운로드 하는 방법은 대표적으로 두가지로 나뉘는데요.
1. tidyverse 를 설치하는 방법
tidyverse 는 datascience를 위해 제작된 R 패키지들의 모음집이라고 할 수 있습니다.
tidyverse 가 포함하는 패키지들로는 : dplyr, ggplot2, tidyr, readr, purrr, tibble, stringr, forcats 등이 있습니다.
따라서, tidyverse 패키지를 다운로드 받음으로써 자연스럽게 dplyr 패키지를 다운받을 수 있게 되는것이죠.
install.packages("tidyverse")
1. dplyr 을 직접 설치하는 방법
혹은 직접적으로 dplyr 패키지를 설치할 수 있습니다.
데이터 전처리만 집중적으로 하고자 하는 경우, dplyr 패키지만 사용해도 무관합니다.
데이터 전처리를 한 후, 이를 그래프로 표현하고자 하는 경우에는 , tidyverse 를 다운받음으로써, dplyr과 ggplot 패키지를 한번에 다운받을 수 있게 되겠죠?
install.packages("dplyr")
<dplyr basics>
dplyr 패키지는 데이터 전처리에 유용한 동사들을 제공합니다.
그중에서 dplyr 틀을 이루는 주요 동사들로는, select(), arrange(), filter(), mutate(), summary() 가 있습니다.
이번 포스팅에서는, dplyr 내장 함수들에 대한 간략하고 핵심적인 기능을 설명해보도록 하겠습니다.
이 내장 함수들을 적절히 설명하기 위해 ggplot2에 내장데이터인 'msleep'을 사용하도록 하겠습니다.
< select() : picks variables based on their names >
select()는 데이터 프레임에서 당신이 관심있어 하는, 즉 분석하고자 하는 Variable만 추출해주는 동사입니다.
dplyr 패키지가 주로 받아서 처리하는 데이터 프레임(data frame)은, 열과 행으로 구성된 2차원의 형태를 가진 데이터 입니다.
데이터 프레임에서는, 열(column)이 변수(variable)을 나타내고, 행(row)은 사례(case)를 나타내죠.
즉, select()함수는, 데이터 프레임에서 분석하고자 하는 열을 추출함으로써, 빠르고 쉽게 데이터의 부분집합에 접근 할 수 있게 만들어주는 함수입니다.
select()함수를 이용해 msleep 데이터에서 열을 추출해봅시다.
1. 하나의 열 추출
#way 1 : set column name
select(msleep, sleep_total) #sleep total 열이 추출됨
#way2 2 : set column number
select(msleep, 1) #첫번째 열(name)이 추출됨
* View()를 통해 select 함수로 열을 추출하여 데이터가 어떻게 변화했는지 직접 확인 가능하십니다! :View(select(msleep, 1))
* str()를 통해 추출된 열에 대한 정보를 얻을 수 있습니다. : str(select(msleep , 1))
1. 두개 이상의 열 추출
두개 이상의 열을 추출하는 방법도 많이 다를 것 없습니다. 그저 data frame 뒤에 추출하고자 하는 열들을 나열하면 됩니다.
#<way 1: set column names>
select(msleep, sleep_total, sleep_rem, brainwt)
#<way 2: set column numbers>
select(msleep, 1, 3, 5)
#<way 3: by using colon>
select(msleep, 1: 3) #첫번째 열부터 세번째 열까지
select(msleep, sleep_total : brainwt) #sleep_total 열부터 brainwt 열까지
3. 선택한 열들을 빼고 추출 : select(data frame, -column names)
특정 열들을 뺀 나머지 열들을 추출하고 싶을때, 빼고싶은 열들 앞에 -를 붙여줌으로써 나머지 열들을 추출할 수 있습니다.
select(msleep, -sleep_total) #sleep_total 열 빼기
select(msleep, -1) #첫번째 열 빼기
select(msleep, -(sleep_total : brainwt)) #sleep_total 부터 brainwt 까지의 열 빼기 < arrange() : changes the ordering of the rows >
arrange()는 데이터를 나열하고자 기준으로 삼을 열(column) 이름을 받아, 행(row)을 재배치 하는 동사입니다.
arrange(data frame, column names) : input으로는 select 함수와 마찬가지로 데이터 프레임과 열 이름을 받습니다.
arrange()함수를 사용하면, input으로 받은 열들을 기준으로, 오름차순(ascending order) 혹은 내림차순 (descending order)로 행이 재배열된 데이터를 보실 수 있습니다.
*특정 열을 기준으로 행을 내림차순으로 나열하고자 할때는, 열을 desc()함수 안에 저장하시면 됩니다.
1. 하나의 열을 기준으로 행 재배치 : 오름차순(ascending order )
*열이 문자형(character)일때는, a 부터 z까지의 알파벳 순서로 행이 나열됩니다.
*열이 숫자형(numeric)일때는, 작은수부터 큰수 순으로 행이 나열됩니다.
arrange(msleep, vore) #vore 열을 기준으로 행 재배열
>View( arrange( msleep, sleep_total )) : sleep_total열을 기준으로 행이 오름차순으로 재배열된 데이터

1. 하나의 열을 기준으로 행 재배치 : 내림차순( descending order )
오름차순(ascending order)로 행을 재배치 할때는, 열 이름을 desc()함수에 넣어서 저장해주셔야 합니다.
*열이 문자형(character)일때는, z 부터 a 까지의 알파벳 역순으로 행이 나열됩니다.
*열이 숫자형(numeric)일때는, 큰수부터 작은수 순으로 행이 나열됩니다.
arrange(msleep, desc(vore)) #vore 열을 기준으로 행을 descending order로 재배열
>View( arrange( msleep, desc(sleep_total ))) : sleep_total열을 기준으로 행이 내림차순으로 재배열된 데이터 입니다.
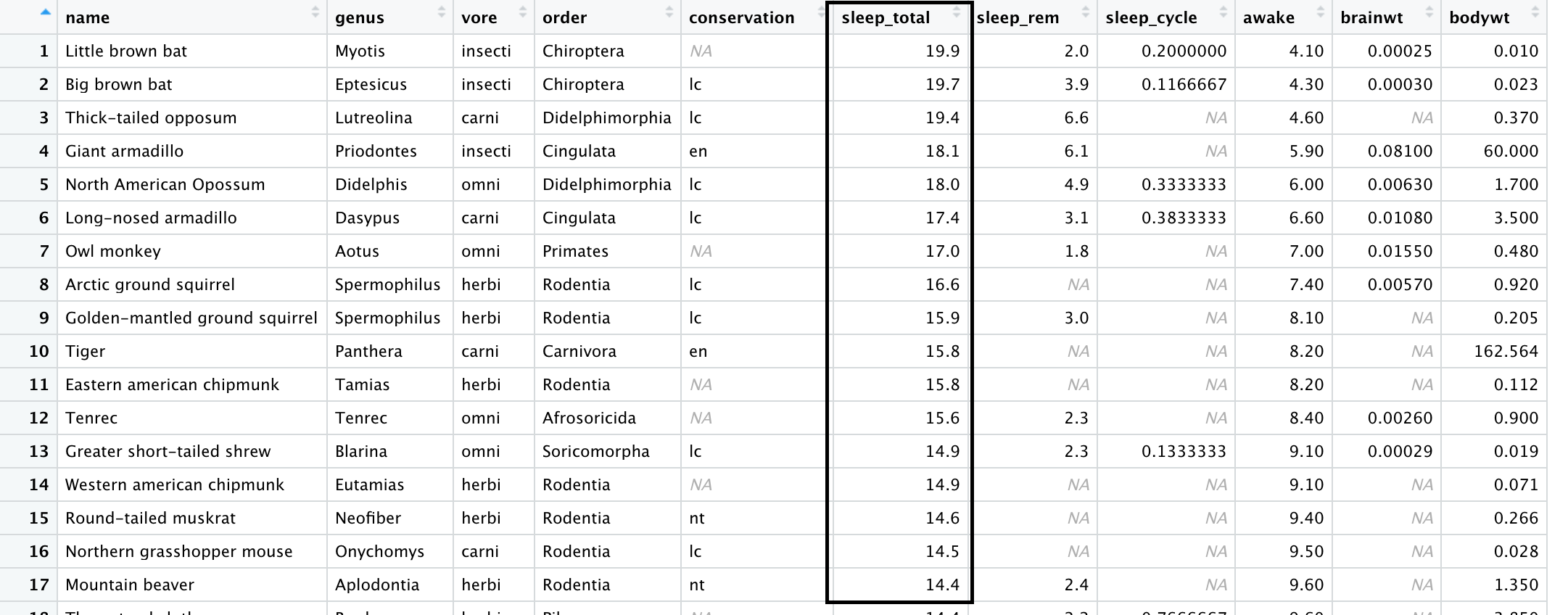
3. 두개 이상의 열을 기준으로 행 재배치
두개 이상의 열을 기준으로 행을 재배치 할때는, data frame 이후에 기준으로 저장하고 싶은 열들을 나열하시면 됩니다.
단, 먼저 지정된 열을 기준으로 행들이 최우선으로 나열된다는 점, 기억해 주세요.
arrange(msleep, vore, desc(sleep_total)) #vore 을 기준으로 행을 오름차순으로 나열 한 후, sleep_total을 기준으로 내림차순 나열
>View(arrange(msleep, vore, desc(sleep_total))
vore 을 기준으로 알파벳 순으로 데이터를 나열한 후, sleep_total을 기준으로 내림차순으로 나열된 모습을 보실 수 있습니다.

4. NA?
arrange()함수를 이용하여 특정 열을 기준으로 오름차순 혹은 내림차순으로 재배열 했을때, NA는 어떻게 처리될까요?
지정된 열에 대해, 행이 NA를 가지는 경우, 이 행들은 맨 마직막에 배열됩니다.

< filter() : picks cases based on their variables >
filter()는 각각의 변수에 대해 주어진 조건에 부합하는 행들을 추출하는 동사입니다.
filter(data frame, 변수에 대한 조건) : input으로는 데이터 프레임과 변수에 대한 조건을 받습니다.
filter()함수에서는 comparison operators(비교 연산자) 와 logical operator (논리 연산자)를 이용해 변수에 대한 조건들을 설정하곤 합니다.
곧 비교 연산자와 논리 연산자에 대한 자세한 설명을 포스팅하겠습니다 :)
간략하게 설명드리자면,
비교 연산자 : >, <, >=, <=, !=, ==
논리 연산자 : & (and), | (or), ! (not)
filter(msleep, vore %in% c("carni", "omni")) #vore 변수가 cardi 혹은 omni인 데이터 추출
table(filter(msleep, vore %in% c("carni", "omni")) $ vore)
#carni omni
# 19 20
filter(msleep, vore == "carni" | vore == "omni") #vore 변수가 cardi 혹은 omni인 데이터 추출
table(filter(msleep, vore == "carni" | vore == "omni")$ vore)
#carni omni
# 19 20
mean(msleep$sleep_total) #10.43373
filter(msleep, vore=="carni", sleep_total < mean(sleep_total)) #vore 변수가 cardi 이면서, sleep_total이 10.43373 보다 작은 행
> View(filter(msleep, vore=="carni", sleep_total < mean(sleep_total)))
: vore 변수는 carni 이면서, sleep_total 변수의 평균보다 작은 sleep_total을 가지고 있는 행들만 추출된 모습을 보실 수 있습니다.

< mutate() : add new variables that are functions of existing variables >
mutate()는 데이터 프레임의 마지막 열에 새로운 변수를 추가해주는 동사입니다.
이때 데이터 프레임에 생긴 새로운 변수는, 먼저 존재하던 변수들에 대한 함수인것이죠.
mutate(data frame, 새로 설정할 변수 이름 = 기존 변수들의 함수)
#하나의 새로운 변수 생성
mutate(msleep, ratio_ram = sleep_rem/sleep_total) #기존에 존재하던 두개의 변수에 대한 함수 : sleep_rem의 sleep_total에 대한 비율
#두개 이상의 새로운 변수 생성
mutate(msleep, ratio_ram = sleep_rem/sleep_total, brain_ratio = brainwt/bodywt)
>View(msleep)
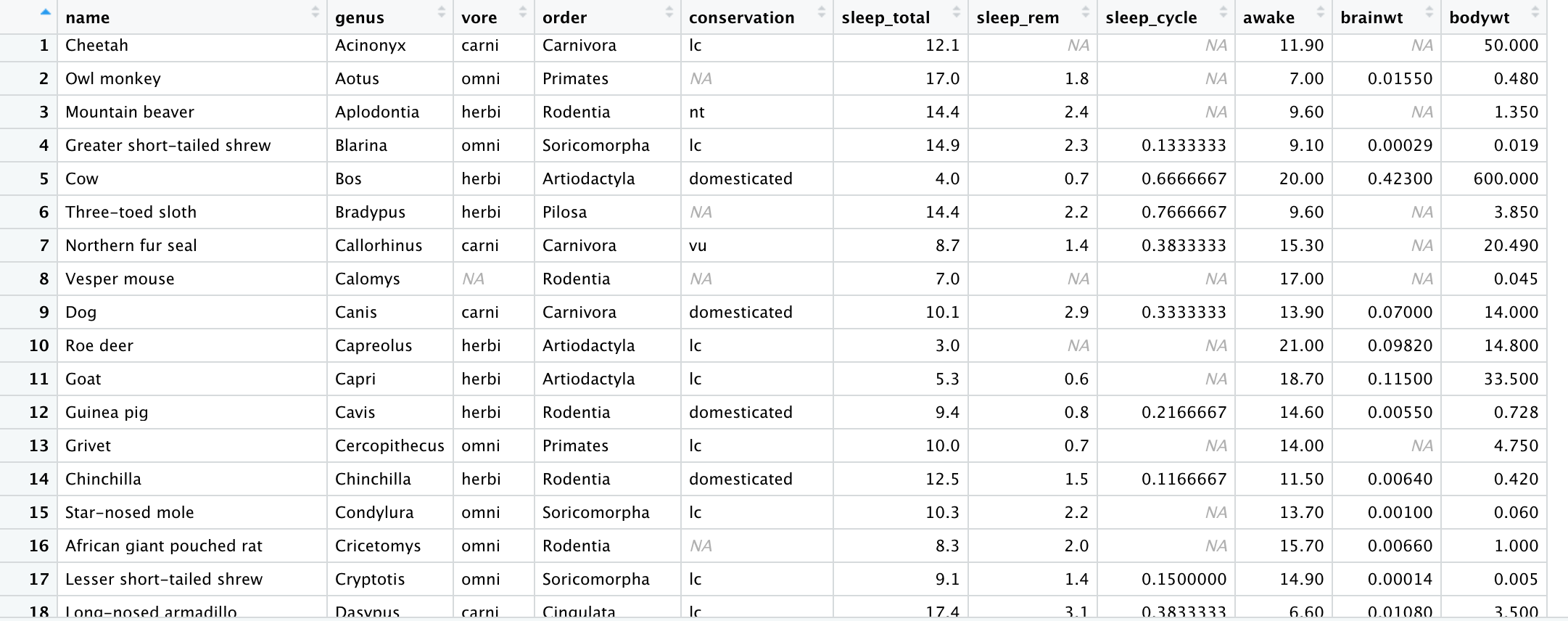
>View(mutate(msleep, ratio_ram = sleep_rem/sleep_total, brain_ratio = brainwt/bodywt))
기존의 변수인 sleep_rem 과 sleep_total 에 대한 새로운 함수인 ratio_remd 변수와,
기존의 변수인 brainwt 와 bodywt 에 대한 새로운 함수인 brain_ratio 가 데이터프레임의 새로운 열로 추가되신것을 보실 수 있습니다.

< transmute() >
mutate() 가 기존 변수들에 대한 함수로 표현되는 새로운 변수를 기존의 데이터 프레임의 마지막열에 추가하는 동사라면,
summarie()는 기존 변수들에 대한 함수로 표현되는 새로운 변수만을 포함하는 데이터 프레임을 만들어주는 동사입니다.
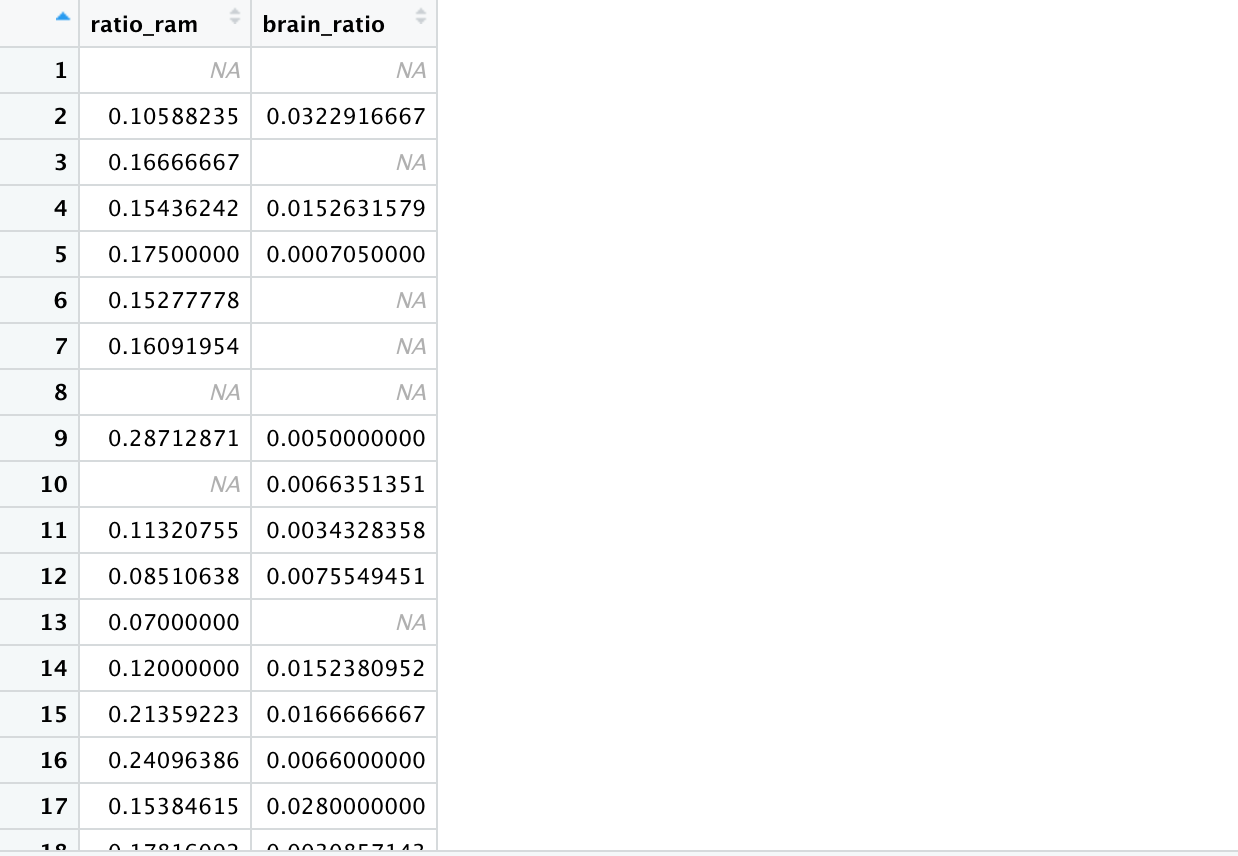
위의 예시에서, mutate아닌, transmute를 사용한다면 다음과 같은 데이터가 생성됩니다.
>View(transmute(msleep, ratio_ram = sleep_rem/sleep_total, brain_ratio = brainwt/bodywt))
< summarise() : reduces multiple variables down to a single summary >
summarise()는 그룹별로 지정된 함수를 적용하여 각 그룹의 특징을 나타내어주는 동사입니다.
summarise()는 주로 group_by()와 함께 쓰입니다. 즉, group_by()함수로 먼저 데이터를 그룹으로 나누어준 후, summarise()에 지정된 함수식이 각 그룹별로 적용되어 나타내어지는 것이죠.
group_by()와 summarise()의 조합은, dplyr패키지에서 가장 유용히 사용되는 조합이므로, 꼭 기억해주세요:)
<하나의 변수에 대해 group으로 나눈 후, summarise>
#<하나의 변수에 대해 group으로 나누어준 후 하나의 함수로 summarise 하기>
msleep_group <- group_by(msleep, vore) # 똑같은 vore 변수값을 가지는 행들끼리 묶어줌
summarise(msleep_group, count = n()) # 각각의 vore에 몇개씩의 데이터가 존재하는지 요약해줌
## A tibble: 5 x 2
# vore count
# <chr> <int>
#1 carni 19
#2 herbi 32
#3 insecti 5
#4 omni 20
#5 NA 7
#<하나의 변수에 대해 group으로 나누어준 후 두개 이상의 함수로 summarise 하기>
msleep_group <- group_by(msleep, vore)
summarise(msleep_group, count = n(), mean_rem = mean(sleep_rem, na.rm = TRUE), sd_rem = sd(sleep_rem, na.rm =TRUE))
## A tibble: 5 x 4
# vore count mean_rem sd_rem
# <chr> <int> <dbl> <dbl>
#1 carni 19 2.29 1.86
#2 herbi 32 1.37 0.922
#3 insecti 5 3.52 1.93
#4 omni 20 1.96 1.01
#5 NA 7 1.88 0.847
<두개 이상의 변수에 대해 group으로 나눈 후, summarise>
#<두개 이상의 변수에 대해 그룹으로 나누어준 후, 한개의 함수로 summarise>
msleep_group <- group_by(msleep, vore, order)
summarise(msleep_group, count = n())
#`summarise()` has grouped output by 'vore'. You can override using the `.groups` argument.
## A tibble: 32 x 3
## Groups: vore [5]
# vore order count
# <chr> <chr> <int>
# 1 carni Carnivora 12
# 2 carni Cetacea 3
# 3 carni Cingulata 1
# 4 carni Didelphimorphia 1
# 5 carni Primates 1
# 6 carni Rodentia 1
# 7 herbi Artiodactyla 5
# 8 herbi Diprotodontia 1
# 9 herbi Hyracoidea 2
#10 herbi Lagomorpha 1
## … with 22 more rows
#<두개 이상의 변수에 대해 그룹으로 나누어준 후, 두개 이상의 함수로 summarise>
summarise(msleep_group, count = n(), mean_rem = mean(sleep_rem, na.rm = TRUE), sd_rem = sd(sleep_rem, na.rm =TRUE))
`summarise()` has grouped output by 'vore'. You can override using the `.groups` argument.
## A tibble: 32 x 5
## Groups: vore [5]
# vore order count mean_rem sd_rem
# <chr> <chr> <int> <dbl> <dbl>
# 1 carni Carnivora 12 1.87 0.996
# 2 carni Cetacea 3 0.1 NA
# 3 carni Cingulata 1 3.1 NA
# 4 carni Didelphimorphia 1 6.6 NA
# 5 carni Primates 1 NaN NA
# 6 carni Rodentia 1 NaN NA
# 7 herbi Artiodactyla 5 0.575 0.126
# 8 herbi Diprotodontia 1 1.5 NA
# 9 herbi Hyracoidea 2 0.55 0.0707
#10 herbi Lagomorpha 1 0.9 NA
## … with 22 more rows여러분이 관심있는 변수를 그룹으로 나누어 각각의 그룹에 대한 요약값들을 이렇게 쉽게 나타낼 수 있다는것이 신기하지 않으신가요? ㅎㅎ
< %>% - chain operator >
위에서, 간략한 설명과 예시를 통해 dplyr 패키지에 존재하는 동사들에 대해 이해가 되셨다면, 마지막으로 이 동사들을 효율적으로 쓰기 위해 필수적인 chain operator 을 설명해드림으로써 오늘의 포스팅을 마무리 하겠습니다.
%>% 로 표현되는 Chain Operator은, dplyr 함수들을 엮어서 데이터 전처리를 효율적으로 수행시켜 줍니다.
위에서는, 각각의 함수가 어떻게 작동하게 되는지 간단하게 설명드렸지만, 실제로 우리가 데이터를 조작하고 분석하기 위해서는, dplyr 패키지에 존재하는 동사들을 엮어서 이용하시게 됩니다.
물론, %>% 를 사용하지 않더라도 괄호를 이용하여 동사들을 연결하거나, 중간결과를 데이터로 저장한 후에 그 데이터를 받아와 새로운 함수를 적용시키는 방식으로도 함수를 엮어서 사용할 수있죠. 하지만, %>% 는 각각의 동사들을 '효율적으로' 묶어 데이터 전처리의 시간과 비용을 줄여줍니다.
%>% 를 쓸때는, 데이터와 그에 적용되는 함수를 순서대로 나열함으로써 복잡한 데이터 전처리를 깔끔하게 나타낼 수 있습니다.
아래의 예시에서는 코드가 간단하고 쉽기때문에, 괄호로 연결하는 경우가 더 깔끔해보인다고 생각이 들 수 있지만,
조금이라도 데이터 전처리 과정이 복잡하고 길어질때는 괄호를 이용해 함수를 이어줄때 많은 실수가 유도될수 있습니다.
summary(head(msleep))
# %>% 를 이용해서 각각의 함수 엮어주기
msleep %>% head() %>% summary()
msleep 데이터를 이용해 복잡한 예시를 들어봅시다.
"msleep 데이터에서 vore, sleep_tal, sleep_rem, brainwt, bodywt 열을 추출 하세요.
그 후, vore가 omni이거나 herbi 인 행들만을 추출하여, 위 변수들에 대한 함수식인 rem_ratio와 brain_ratio 라는 새로운 변수를 만들어 데이터 프레임에 더해준 후, vore의 알파벳 순으로 데이터를 정리하여 주십시오 " 라는 미션을 들었을때 %>% 를 사용하면 효율적이고 정확한 코드를 작성 할 수 있어요.
괄호를 이용해서 함수들을 이어줄때, 중간결과를 새로운 데이터로 저장한 후 새로운 함수를 적용시킬때, %>% 을 사용할때 이 세가지 케이스를 비교해봅시다.
1. 괄호를 이용 :
-> arrange(mutate(filter(select(msleep, vore, sleep_total, sleep_rem, brainwt, bodywt), vore == "omni" | vore == "herbi"), rem_ratio = sleep_rem/sleep_total, brain_ratio = brainwt/bodywt), vore)
arrange(mutate(filter(select(msleep, vore, sleep_total, sleep_rem, brainwt, bodywt), vore == "omni" | vore == "herbi"), rem_ratio = sleep_rem/sleep_total, brain_ratio = brainwt/bodywt), vore)
## A tibble: 52 x 7
# vore sleep_total sleep_rem brainwt bodywt rem_ratio brain_ratio
# <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
# 1 herbi 14.4 2.4 NA 1.35 0.167 NA
# 2 herbi 4 0.7 0.423 600 0.175 0.000705
# 3 herbi 14.4 2.2 NA 3.85 0.153 NA
# 4 herbi 3 NA 0.0982 14.8 NA 0.00664
# 5 herbi 5.3 0.6 0.115 33.5 0.113 0.00343
# 6 herbi 9.4 0.8 0.0055 0.728 0.0851 0.00755
# 7 herbi 12.5 1.5 0.0064 0.42 0.12 0.0152
# 8 herbi 5.3 0.5 0.0123 2.95 0.0943 0.00417
# 9 herbi 3.9 NA 4.60 2547 NA 0.00181
#10 herbi 2.9 0.6 0.655 521 0.207 0.00126
## … with 42 more rows우리는 괄호로 동사들을 잘 이어 다행스럽게도 의도한대로 데이터를 추출했다고 하지만, 위의 코드 너무 복잡하지 않나요?
위처럼 괄호를 이용해 각 함수를 연결해주었을때는, 실수를 할 확률도 클 뿐 아니라, 어느 부분에서 에러가 났는지 캐치하기 너무 복잡한 코드가 만들어집니다.
2. 새로운 객체 생성 후 함수 적용 :
그렇다고 각각의 단계에 새로운 객체명을 지정해주고 새로 만들어진 데이터를 반복적으로 새로운 동사에 적용시킨다고 했을때는 코드가 너무 비효율적으로 짜여졌다는 생각이 듭니다.
msleep_1 <- select(msleep, vore, sleep_total, sleep_rem, brainwt, bodywt)
msleep_2 <- filter(msleep_1, vore == "omni" | vore == "herbi")
msleep_3 <- mutate(msleep_2, rem_ratio = sleep_rem/sleep_total, brain_ratio = brainwt/bodywt)
msleep_4 <- arrange(msleep_3, vore)
%>% : chain operator
이제야 비로소, %>%의 효율성과 안정성을 설명할 수 있겠네요.
각각의 코드를 작성 한 후, %>%로 함수들을 이어주는 법을 위의 예시를 사용하여 표현해볼게요.
msleep %>% select(vore, sleep_total, sleep_rem, brainwt, bodywt) %>% filter(vore == "omni" | vore == "herbi") %>% mutate(rem_ratio = sleep_rem/sleep_total, brain_ratio = brainwt/bodywt)%>%arrange(vore)
## A tibble: 52 x 7
# vore sleep_total sleep_rem brainwt bodywt rem_ratio brain_ratio
# <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
# 1 herbi 14.4 2.4 NA 1.35 0.167 NA
# 2 herbi 4 0.7 0.423 600 0.175 0.000705
# 3 herbi 14.4 2.2 NA 3.85 0.153 NA
# 4 herbi 3 NA 0.0982 14.8 NA 0.00664
# 5 herbi 5.3 0.6 0.115 33.5 0.113 0.00343
# 6 herbi 9.4 0.8 0.0055 0.728 0.0851 0.00755
# 7 herbi 12.5 1.5 0.0064 0.42 0.12 0.0152
# 8 herbi 5.3 0.5 0.0123 2.95 0.0943 0.00417
# 9 herbi 3.9 NA 4.60 2547 NA 0.00181
#10 herbi 2.9 0.6 0.655 521 0.207 0.00126
## … with 42 more rowschain operator 을 사용할때는, 분석의 대상이 되는 데이터를 맨 앞에 두고 %>% 로 적용할 함수 순으로 코드를 작성하면 됩니다.
위에서 dply에 존재하는 함수의 기능을 설명할때는, 각각의 함수가 데이터를 input으로 직접 받았습니다.
>transmute(msleep, ratio_ram = sleep_rem/sleep_total, brain_ratio = brainwt/bodywt)
%>% 을 사용할때는, 각 함수가 input으로서 데이터 프레임을 포함하지 않아도 됩니다. 데이터 프레임 이외의 input들은 그대로 써주시면 되어요.
>msleep %>% transmute(ratio_ram = sleep_rem/sleep_total, brain_ratio = brainwt/bodywt)
드디어!! dplyr basics라고 불리우는 dplyr의 주요 함수들에 대한 설명을 간략하게 했는데요.
다음 포스팅에서는 각각의 동사들에 같이 사용되면 좋은 함수들을 자세하게 설명해볼게요 :)
오늘도 봐주셔서 감사합니당:)
ref.
https://dplyr.tidyverse.org/reference/index.html#section-vector-functions
https://r4ds.had.co.nz/transform.html#dplyr-basics
https://www.tidyverse.org/packages/
'R' 카테고리의 다른 글
| [회귀] 범주형 독립변수를 지시변수로 포함한 선형 회귀모형/교호작용 / R (0) | 2021.11.02 |
|---|---|
| (R) 비교연산자, 논리연산자 / filter() in dplyr / near(), between(), %in% (0) | 2021.06.13 |
| (R) apply 계열 함수/apply, lapply, sapply, mapply, tapply / 함수 동시 적용/ 예시 (0) | 2021.06.03 |
| 확률적 표본 추출법 R/ 단순 임의 추출, 체계적 추출, 층화 임의 추출, 군집 추출 (0) | 2021.05.26 |
| R 데이터 구조와 색인(Indexing)/ scalar, vector, factor, matrix, array, data frame, list (0) | 2021.05.16 |

